Yuheng Luo, một học viên cao học tại Đại học Hawaii ở Mnoa, đã phát hiện ra trục trặc khi cố gắng xác minh kết quả nghiên cứu của giáo sư hóa học Philip Williams về vi khuẩn lam. Luo đã sử dụng một kịch bản (script) viết bằng Python trong bài báo công bố năm 2014 trên Nature Protocol.
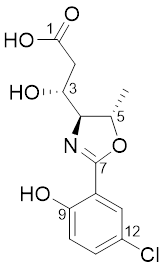
Với cùng một kịch bản trên, các nhà nghiên cứu đã rất ngạc nhiên khi thấy kết quả tính toán của họ thay đổi tùy thuộc vào hệ điều hành được sử dụng, gây ra sự khác biệt giữa các hệ điều hành Mac, Windows và Linux. Chẳng hạn, tính toán giá trị độ chuyển dịch hóa học của cacbon trong hợp chất leptazoline A rất khác nhau chạy trên các hệ điều hành Mac, Windows và Linux (Bảng 1). Trong đó, kết quả tính toán trên LINUX (Ubuntu16), Mac (Mojave) không chính xác.
Bảng 1. Giá trị tính toán độ chuyển dịch hóa học của hợp chất leptazoline A
|
leptazoline A |
C |
LINUX (Ubuntu16) |
Windows (ver. 10) |
Mac (Mavericks) |
Mac (Mojave) |
|
1 |
172.4 |
173.2 |
173.2 |
172.7 |
|
|
2 |
36.0 |
37.7 |
37.7 |
39.3 |
|
|
3 |
68.3 |
68.4 |
68.4 |
69.0 |
|
|
4 |
70.6 |
70.5 |
70.5 |
71.2 |
|
|
5 |
79.0 |
78.4 |
78.4 |
79.0 |
|
|
6 |
15.5 |
13.3 |
13.3 |
13.3 |
|
|
7 |
162.4 |
162.5 |
162.5 |
161.8 |
|
|
8 |
110.4 |
109.8 |
109.8 |
110.3 |
|
|
9 |
155.0 |
156.5 |
156.5 |
155.5 |
|
|
10 |
116.2 |
115.5 |
115.5 |
116.0 |
|
|
11 |
131.6 |
131.6 |
131.6 |
131.7 |
|
|
12 |
127.1 |
126.6 |
126.6 |
127.0 |
|
|
13 |
126.7 |
125.6 |
125.6 |
126.3 |
Sự sai lệch có thể gây ra những sai lầm nghiêm trọng trong nghiên cứu cấu trúc của hợp chất tự nhiên (đặc biệt là xác định hóa lập thể), xác định sản phẩm của các phản ứng hóa học, tìm hiểu con đường sinh tổng hợp… Các nhà nghiên cứu mô tả phát hiện của họ và xuất bản một phiên bản sửa lỗi với khoảng 1.000 dòng mã. Công trình này được công bố trên tạp chí Organic Letters (DOI: 10.1021/acs.orglett.9b03216).
Hiện vẫn chưa rõ có bao nhiêu công trình đã xuất bản bị ảnh hưởng bởi lỗi này. Vì đa phần các tác giả thường không đề cập đến hệ điều hành sử dụng trong bài báo của họ. Theo số liệu từ Nature Protocol, bài báo gốc năm 2014 đã được truy cập gần 1.900 lần và được trích dẫn trong 158 nghiên cứu khác. Phát hiện lỗi lần này khuyến cáo các nhà nghiên cứu nên kiểm tra lại kết luận trong các công trình đã công bố.
Tổng hợp
TS. Hồ Việt Đức
Tham khảo:
- Bhandari Neupane, J., Neupane, R. P., Luo, Y., Yoshida, W. Y., Sun, R., & Williams, P. G. (2019). Characterization of Leptazolines A–D, polar oxazolines from the cyanobacterium Leptolyngbya sp., Reveals a glitch with the “Willoughby–Hoye” scripts for calculating NMR chemical shifts. Organic Letters, 21(20), 8449-8453. doi: 10.1021/acs.orglett.9b03216.
- Willoughby, P. H.; Jansma, M. J.; Hoye, T. R. (2014), A guide to smallmolecule structure assignment through computation of (1H and 13C) NMR chemical shifts. Protoc., 9, 643-660.
- Thử nghiệm phi động vật – Kỷ nguyên mới cho nghiên cứu phát triển thuốc
- Qfitlia – phương pháp điều trị mới cho Hemophilia A hoặc B
- Mô hình “Netflix plus”: Giải pháp tài chính đột phá cải thiện tiếp cận thuốc tại các nước thu nhập thấp và trung bình
- Tỷ lệ Cholesterol “Xấu” (Non-HDL) trên Cholesterol “Tốt” (HDL) Cao Liên Quan Đến Nguy Cơ Tăng Huyết Áp và Bệnh Tim Mạch
- 6 loại thảo dược có khả năng gây tổn thương gan
- Tirzepatide giúp cải thiện tình trạng suy tim phân suất tống máu bảo tồn trên bệnh nhân béo phì
- FDA chấp thuận Ebglyss (Lebrikizumab) điều trị viêm da dị ứng
- Bệnh Alzheimer: Các loại thuốc mới được chấp thuận trên lâm sàng có tạo ra sự khác biệt thực sự không?
- FDA chấp thuận vắc-xin cúm dạng xịt mũi đầu tiên sử dụng tại nhà
- Tổn thương gan do thuốc
- Bốn hợp chất phenolic mới từ quả của loài Alpinia galanga
- Tác dụng kháng virus của gamma-mangostin
- Chế độ ăn Keto có thể đẩy nhanh quá trình lão hóa cơ quan
- Một số điểm mới trong quy định giá dịch vụ khám, chữa bệnh bảo hiểm y tế giữa các bệnh viện cùng hạng trên toàn quốc từ 17/11/2023
- Si rô ngô giàu fructose thúc đẩy sự phát triển khối u đường ruột ở chuột
- FDA cấp phép phê duyệt nhanh AMTAGVI điều trị ung thư hắc tố da