Hiện nay, sự đề kháng kháng sinh đã và đang trở thành một hiểm họa đáng báo động đối với sức khỏe của con người. Nếu không được quan tâm đúng mức, các nhà khoa học dự báo số ca tử vong sẽ lên đến 10 triệu và gây thiệt hại cho nền kinh tế lên đến 1 ngàn tỉ đô la vào năm 2050. Để đối phó với tình trạng đề kháng, thuận tiện trong theo dõi cũng như đảm bảo kết quả điều trị tốt nhất cho bệnh nhân, chuyên gia y tế cần có phương pháp có thể xác định nhanh tính nhạy cảm kháng sinh (test nhạy cảm kháng sinh, TNKS), và lý tưởng nhất là thực hiện được TNKS ngay tại lúc thăm khám (point of care) thay vì các phương pháp xét nghiệm truyền thống cần tốn thời gian vài giờ đến vài ngày sau khi lấy mẫu. Nhu cầu này cũng đã được nêu lên trong các hướng dẫn điều trị bằng kháng sinh bởi Trung tâm Dự phòng và Kiểm soát Bệnh tật Hoa Kỳ (CDC) và Tổ chức Y tế Thế giới (WHO).
Một ví dụ điển hình cho tính cấp thiết của TNKS nhanh là trường hợp nhiễm khuẩn đường tiết niệu (NKTN), một trong những loại nhiễm khuẩn phổ biến nhất với khoảng 8 triệu ca mỗi năm và hầu như luôn sử dụng kháng sinh để điều trị. Do chưa có TNKS nhanh, bác sĩ thường dùng các loại kháng sinh thay thế (second-line) như ciprofloxacin thay vì kháng sinh đầu tay (first-line) như nitrofurantoin để điều trị NKTN. Hệ quả là làm tăng mức độ sử dụng và tỉ lệ đề kháng của vi khuẩn đối với ciprofloxacin, làm hạn chế các lựa chọn trong điều trị, đặc biệt là trong các tình huống nghiêm trọng khi NKTN tiến triển sang nhiễm khuẩn huyết. Như vậy, việc rút ngắn thời gian xác định mẫu vi khuẩn có đề kháng đối với kháng sinh hay không sẽ giúp quá trình điều trị nhiễm khuẩn được tiến hành nhanh chóng và hiệu quả.
Do đó, tác giả Schoepp, Schlappi và cộng sự (2017) đã phát triển một test nhạy cảm kháng sinh có thể tiến hành chỉ trong vòng 30 phút với mẫu nước tiểu của bệnh nhân. Test này sử dụng phương pháp số hóa khuếch đại đẳng nhiệt bằng đoạn vòng (Digital loop-mediated isothermal amplification, dLAMP), qua đó có thể định lượng các nucleic acid maker chỉ điểm cho tính nhạy cảm đối với kháng sinh. Các marker này được sinh ra sau khi vi khuẩn trong mẫu xét nghiệm được ủ với kháng sinh trong thời gian ngắn. Các marker được khuếch đại và định lượng bằng một thiết bị đặc biệt và nhỏ gọn gọi là SlipChip, trong đó việc định lượng được theo dõi ở thời gian thực và hoàn toàn số hóa.
TNKS nhanh gồm hai tiến trình cơ bản: cho tiếp xúc với kháng sinh và định lượng marker nhạy cảm kháng sinh. Quy trình thực hiện gồm các bước: chia đều và pha loãng mẫu nước tiểu thành hai phần có thể tích bằng nhau – mẫu thử có kháng sinh và mẫu đối chứng không có kháng sinh. Sau khi ủ khoảng 15 phút, hai mẫu được định lượng chuỗi nucleic acid đích (marker TNKS) và tính tỉ lệ nồng độ marker giữa mẫu chứng và mẫu thử (control/treated, CT). So sánh giá trị CT này với giá trị ngưỡng CT xác định trước, nếu cao hơn ngưỡng thì kết luận vi khuẩn trong mẫu xét nghiệm nhạy cảm kháng sinh, thấp hơn thì kết luận là đề kháng.
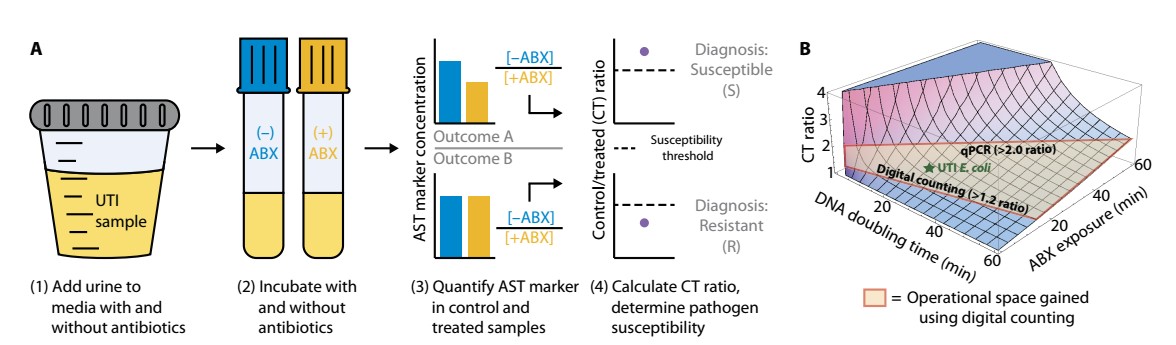
Hình 1. (A) Tiến trình thực hiện TNKS nhanh: 1. Cho mẫu nước tiểu vào môi trường không có/có kháng sinh (mẫu chứng/mẫu thử) 2. Ủ hai mẫu 3. Định lượng marker TNKS trong hai mẫu 4. Tính tỉ lệ CT, xác định tính nhạy cảm của vi khuẩn. (B) Mô hình lý thuyết dự đoán tỉ lệ CT phụ thuộc vào thời gian nhân đôi DNA của vi khuẩn và thời gian tiếp xúc với kháng sinh.
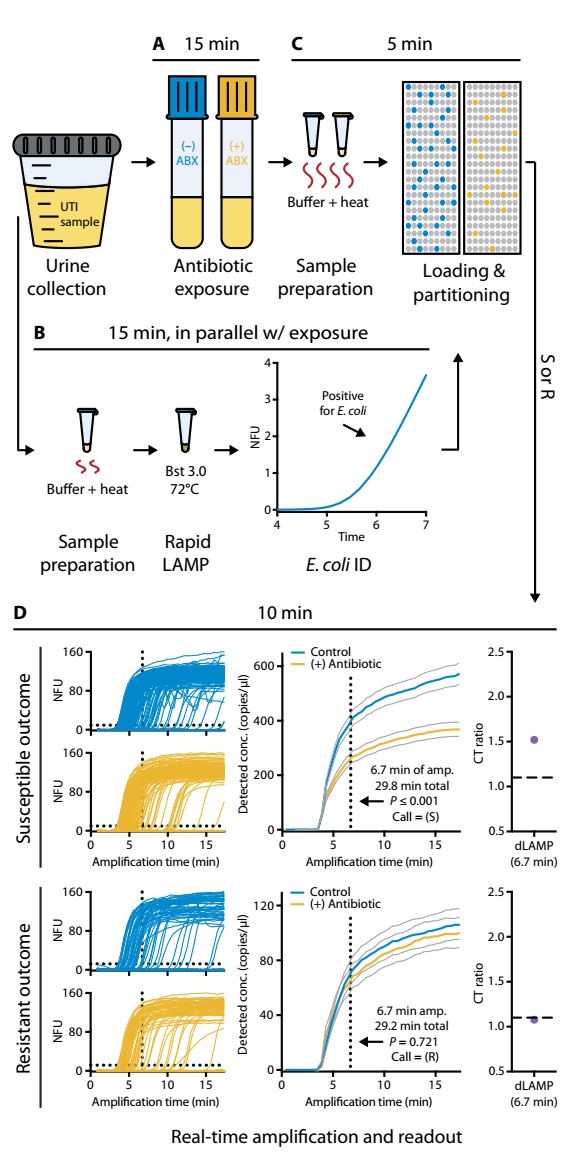
Hình 2. Mô tả quy trình thực nghiệm trong 30 phút (từ khi có mẫu đến lúc cho kết quả nhạy cảm kháng sinh) (A) Mẫu nước tiểu của bệnh nhân có nhiễm khuẩn niệu được cho vào môi trường có (màu vàng) và không có kháng sinh (màu xanh), ủ trong 15 phút. (B) Test LAMP được thực hiện song song với một mẫu nước tiểu khác, cho thấy sự có mặt của vi khuẩn E. coli. (C) Phép định lượng nucleic acid bằng dLAMP được thực hiện trên SlipChip, trong đó mẫu được phân chia nhanh vào các vi ngăn. (D) Kiểm soát quá trình dLAMP theo thời gian thực, kết quả của mẫu nhạy cảm kháng sinh và đề kháng kháng sinh được thể hiện tương ứng.
Test sử dụng một chip số hóa được gọi là SlipChip để khuếch đại và định lượng marker. Đây là một thiết bị tạo ra vi dòng chảy phân chia mẫu vào 1280 vi ngăn (khoảng 2,5 nL mỗi ngăn) để thực hiện các phản ứng khuếch đại nucleic acid. Nếu có mặt các phân tử đích ở mỗi vi ngăn, chúng sẽ được khuếch đại và tổng số ngăn dương tính sẽ được đếm theo thời gian thực. Với phương pháp vi dòng này, nhóm nghiên cứu đã xác định được độ nhạy cảm kháng sinh của vi khuẩn Escherichia coli nhanh hơn nhiều so với phương pháp kinh điển.
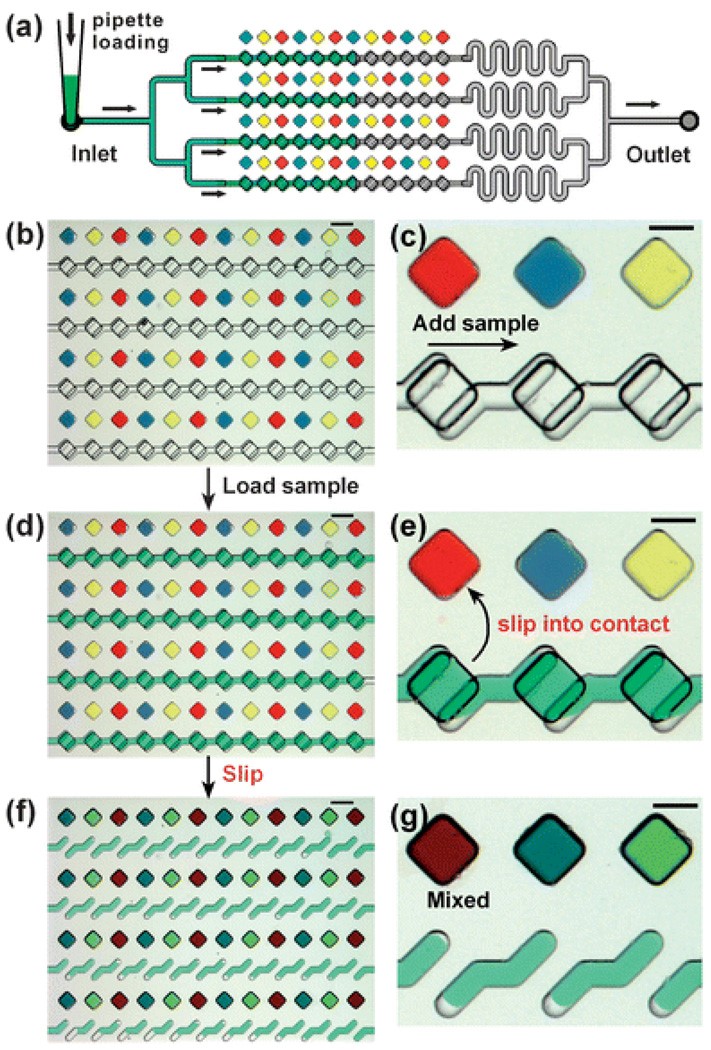
Hình 3. (a) Nguyên lý hoạt động của SlipChip: Chất phản ứng được nhỏ vào đầu inlet, lực mao dẫn giúp dung dịch được chảy dọc theo rãnh định sẵn và phân bố vào các vi ngăn. (b-g) Hình ảnh thực tế của 1 loại SlipChip, sử dụng dung dịch màu trong các vi ngăn để minh họa. Chip gồm có 2 tấm áp vào nhau, khi 2 tấm được dịch chuyển để vi ngăn trên mỗi tấm trùng khít nhau thì các dung dịch được trộn lẫn và phản ứng xảy ra
Phương pháp LAMP được phát triển và công bố lần đầu tiên vào năm 2000 bởi các nhà nghiên cứu của công ty Eiken Chemical Co., Nhật Bản. Đây được xem là phương pháp khuếch đại gen đơn giản, nhanh, đặc hiệu và có hiệu quả kinh tế cao so với phương pháp PCR truyền thống. Phương pháp này sử dụng 4 đoạn mồi được thiết kế đặc hiệu để nhận biết 6 vùng phân biệt trên đoạn gen đích, thực hiện phản ứng thay thế chuỗi tại nhiệt độ hằng định. Việc khuếch đại và nhận biết gen được hoàn thành chỉ qua một bước, bằng cách ủ hỗn hợp mẫu, đoạn mồi, DNA polymerase có hoạt tính thay thế chuỗi, và chất nền ở nhiệt độ cố định khoảng 65 oC. Sau 15-60 phút, DNA được nhân lên 109-1010 lần. Do tính đặc hiệu cao, sự có mặt của sản phẩm được khuếch đại chứng tỏ sự có mặt của gen đích. Phương pháp này có tiềm năng ứng dụng trên nhiều lĩnh vực như xét nghiệp nhanh tại chỗ, test xét nghiệm huyết thống ở những nước đang phát triển với trang bị đơn giản, test nhanh sản phẩm thực phẩm hoặc môi trường. Về sau, nó được cải tiến để việc định lượng được tiến hành một cách số hóa, trở thành phương pháp dLAMP.
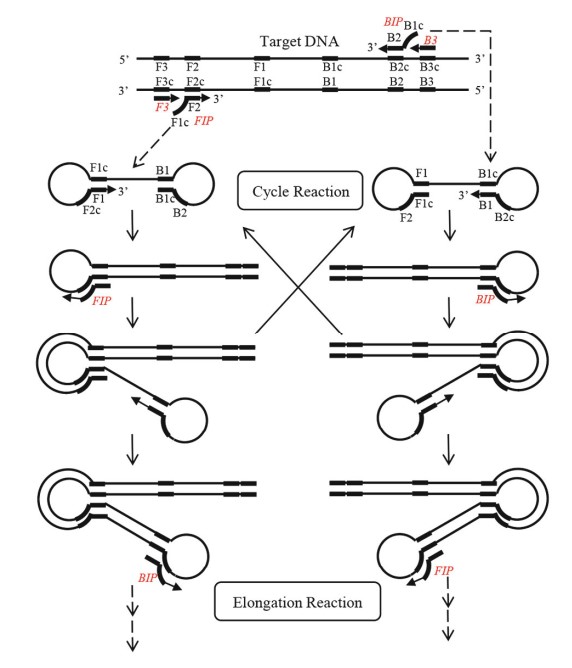
Hình 4. Mô tả quá trình diễn ra trong phương pháp khuếch đại gen LAMP, sau khi đoạn mồi tổng hợp nên các đoạn gen vòng (loop), các gen vòng này khởi động một loạt các chu kỳ tự sao chép sinh ra sản phẩm với số lượng tăng lên rất nhanh trong thời gian ngắn
Lược dịch và tổng hợp
Võ Quốc Hùng
————–
Tài liệu tham khảo:
- (CDC) Centers for Disease Control and Prevention (2013), Antibiotic resistance threats in the United States, (CDC Office of Infectious Diseases, 2013).
- (WHO) World Health Organization (2015), Global antimicrobial resistance surveillance system—Manual for early implementation (WHO, 2015).
- Czajka Caitlin (2017), Rapidly recognizing resistance, Science, 358 (6359), 76-77.
- Schoepp Nathan G., Schlappi Travis S., Curtis Matthew S., Butkovich Slava S., Miller Shelley, Humphries Romney M., Ismagilov Rustem F. (2017), Rapid pathogen-specific phenotypic antibiotic susceptibility testing using digital LAMP quantification in clinical samples, Science Translational Medicine, 9, eaal3693.
- Du Wenbin, Li Liang, Nichols Kevin P., Ismagilov Rustem F. (2009), SlipChip, Lab on a chip, 9 (16), 2286–92.
- Notomi T., Mori Y., Tomita N., Kanda H. (2015), Loop-mediated isothermal amplification (LAMP): principle, features, and future prospects [In eng], J Microbiol, 53 (1), 1-5.
- Lynkuet – thuốc mới điều trị chứng bốc hỏa do mãn kinh
- Thử nghiệm phi động vật – Kỷ nguyên mới cho nghiên cứu phát triển thuốc
- Qfitlia – phương pháp điều trị mới cho Hemophilia A hoặc B
- Mô hình “Netflix plus”: Giải pháp tài chính đột phá cải thiện tiếp cận thuốc tại các nước thu nhập thấp và trung bình
- Tỷ lệ Cholesterol “Xấu” (Non-HDL) trên Cholesterol “Tốt” (HDL) Cao Liên Quan Đến Nguy Cơ Tăng Huyết Áp và Bệnh Tim Mạch
- 6 loại thảo dược có khả năng gây tổn thương gan
- Tirzepatide giúp cải thiện tình trạng suy tim phân suất tống máu bảo tồn trên bệnh nhân béo phì
- FDA chấp thuận Ebglyss (Lebrikizumab) điều trị viêm da dị ứng
- Bệnh Alzheimer: Các loại thuốc mới được chấp thuận trên lâm sàng có tạo ra sự khác biệt thực sự không?
- FDA chấp thuận vắc-xin cúm dạng xịt mũi đầu tiên sử dụng tại nhà
- Tổn thương gan do thuốc
- Bốn hợp chất phenolic mới từ quả của loài Alpinia galanga
- Tác dụng kháng virus của gamma-mangostin
- Chế độ ăn Keto có thể đẩy nhanh quá trình lão hóa cơ quan
- Một số điểm mới trong quy định giá dịch vụ khám, chữa bệnh bảo hiểm y tế giữa các bệnh viện cùng hạng trên toàn quốc từ 17/11/2023
- Si rô ngô giàu fructose thúc đẩy sự phát triển khối u đường ruột ở chuột
